Ces bases de données ont été construites selon les principes FAIR (Findable, Accessible, Interoperable, and Re-usable) en extension des portails Glyco3D (http://glyco3d.cermav.cnrs.fr/ et MatrixDB (http://matrixdb.univ-lyon1.fr/).

EPS-DB est une base de données Web (http://www.epsdatabase.com) consacrée aux exopolysaccharides bactériens (EPS). L’originalité de cette base est de couvrir les structures et les propriétés fonctionnelles. Chaque EPS est caractérisé par ses différents niveaux de structure, sa taxonomie, les conditions de croissance et d’extraction des souches, les propriétés fonctionnelles ainsi que les protéines en interactions. L’utilisation d’une nomenclature et d’un mode de représentation normalisés assure l’interopérabilité avec les bases de données majeures du domaine (doi:10.1093/glycob/cwy084)
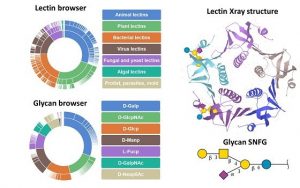
Les lectines (protéines interagissant avec des carbohydrates de manière réversible) jouent un rôle essentiel dans les interactions cellulaires. Utilisées comme outils de criblage, elles constituent également des éléments de constructions d’architectures complexes. UniLectin3D (https://www.unilectin.eu/unilectin3D/) est une base de données ayant fait l’objet d’une curation manuelle. Une classification des lectines selon leurs structures 3D et les ligands avec lesquels elles interagissent confère à cette base de données de nombreuses perspectives d’analyse, de compréhension et de prédiction. UniLectin3D succède au volet lectines du portail glyco3D, et s’accompagne de nombreux liens vers d’autres bases de données: UniProt pour les protéines, PLIP pour les interactions, PCB Care pour la validation de structure des sucres (doi: 10.1093/nar/gky832).
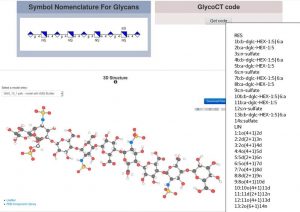
MatrixDB (http://matrixdb.univ-lyon1.fr/) est une base de données d’interactions biomoléculaires focalisée sur la matrice extracellulaire (MEC) et notamment sur les interactions entre les protéines de la matrice extracellulaire et les glycosaminoglycanes (GAG) tels que l’héparine/héparane sulfate, l’acide hyaluronique, le dermatane, le chondroïtine et le kératane sulfate. MatrixDB possède une nouvelle fonctionnalité qui permet désormais de générer des structures 3D à partir d’une séquence de GAG. Cette fonctionnalité fait partie d’un pipeline qui convertit les séquences de GAGs dans des formats standardisés (IUPAC, SNFG et GlycoCT) disponibles dans MatrixDB puis génère les modèles 3D au format PDB qui est compatible avec la majorité des logiciels graphiques (PyMol, VMD, SweetUnityMol ,…) (doi : 10.1093/nar/gky1035 et 10.1093/glycob/cwy084).